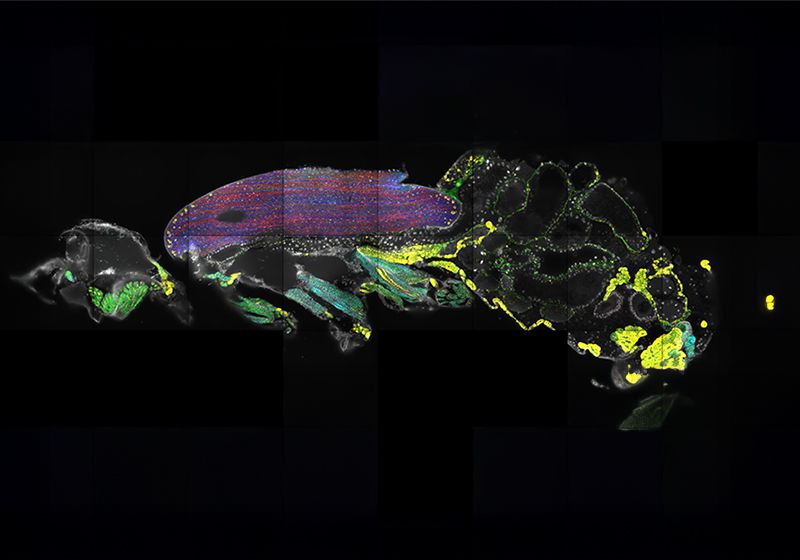
Os cientistas usaram a cartografia molecular para localizar as posições de mRNAs em todo o corpo de uma mosca de frutas de Drosophila.
Frank Snorror / Licença CC-BY 4.0Janssens et al., 2025, eLife
KNo entanto, a localização de um gene dentro do tecido intacto ou de uma única célula permite que os cientistas desbloqueem funções celulares desconhecidas. Esta informação é frequentemente perdido na maioria das técnicas de sequenciamento genético, mas transcriptômica espacial oferece uma maneira de superar essa limitação.1,2
“Se você tem uma máquina – por exemplo, uma bicicleta – todos os diferentes componentes da bicicleta precisariam ser adequados nos lugares certos para que eles funcionassem”, disse Lambda Mosesum biólogo computacional da Universidade de Columbia. “A transcriptômica espacial permite obter a localização desses componentes.”
Em um estudo publicado em eLifeos cientistas usaram transcriptômica espacial e um painel de genes direcionados para capturar as posições de 150 mRNAs em todo o corpo, incluindo o cérebro, de um adulto Drosophila melanogaster.3 Ao fazer isso, eles foram capazes de mapear os locais de tipos e clusters exclusivos e anteriormente não caracterizados. Os resultados acrescentam mais profundidade ao atlas de célula única da mosca, preparando o cenário para futuros estudos mecanicistas.
““Drosophila é como uma faca do exército suíço ”, disse Pierre Mangeolum biofísico na Universidade de Aix-Marseille e co-autor do estudo. Por causa deles genes bem definidos e neurônios isso função como aqueles em mamíferosAssim, Drosophila são proxies valiosos para estudar funções do cérebro humano.4,5
Jasper Janssensum biólogo de desenvolvimento da ETH Zurique e co -autor no estudo, anteriormente sequenciado Mais de 570.000 células na mosca adulta quando ele era um estudante de graduação em Ku Leuven.6 Usando sequenciamento de célula única, ele e sua equipe anotaram 250 desconhecidos Drosophila tipos de células. Apesar desse esforço, milhares de células permaneceram não caracterizadas devido à falta de informações que ligam a transcrição de genes à morfologia da mosca, disse Janssens.
Para abordar essa limitação, os pesquisadores se voltaram para Cartografia molecular-Um método transcriptômico espacial baseado em imagem, baseado em imagens, que permite que os cientistas visualizem moléculas únicas de RNA em tecido intacto.7 A técnica utiliza sondas genéticas marcadas com fluorescência que se ligam a segmentos específicos de ácido ribonucleico. Os cientistas congelam uma mosca de frutas e o cortou em seções de 10 micrômetro ao longo da parte média do corpo, permitindo que eles visualizem a maioria de seus órgãos. Para o estudo de prova de conceito, Janssens e sua equipe usaram um painel de 150 genes-100 mRNAs direcionados expressos no cérebro, enquanto os 50 mRNAs direcionados restantes no corpo.
“É um estudo muito legal”, disse Moses, que não estava envolvido no estudo. ““Com algo como cartografia molecular, podemos ver onde as transcrições ou moléculas de mRNA estão localizadas dentro das células. E muitas vezes isso pode ser muito interessante, porque as transcrições podem ser transportadas para diferentes locais da célula para executar funções biológicas. ” Por exemplo, nos neurônios, o mRNA se move do núcleo para os espinhos dendríticos, onde estão envolvidos na atividade sináptica Localização de mRNA Indica tipos de células distintos, estágios de desenvolvimento e fenótipos de tecido.8
Neste estudo, os cientistas observaram a localização variada do mRNA que codifica a titina – uma proteína gigante abundante no músculo. “Agora descobrimos que os núcleos terminais que estão no final do músculo (de vôo) expressam mais (esses genes)”, disse o co -autor do estudo Frank Snorrerum especialista em dinâmica muscular na Universidade de Aix-Marseille. “Foi completamente inesperado”, acrescentou, porque a proteína da titina é necessária em todo o músculo, não apenas nas bordas. O resultado mostrou que a localização do mRNA e as atividades transcricionais dentro dessas células musculares de vôo são mais diversas do que os cientistas anteriormente pensados e podem apontar para mecanismos distintos que desencadearão investigações futuras.
A equipe agora está buscando maneiras de criar um mapa 3D para identificar genes em células sobrepostas que são difíceis de resolver em 2D.